Une collaboration entre l’Université Rice, le Baylor College of Medicine et le Jan et le Dan Duncan Neurological Research Institute (NRI) du Texas Children Hospital ont produit une percée dans la façon d’étudier et de classer les maladies complexes.
La nouvelle méthode, appelée pivot causal, donne aux chercheurs un moyen puissant de détecter les moteurs génétiques cachés et les patients sous-groupes par les véritables causes biologiques de leurs maladies à l’aide de nouveaux outils de calcul développés dans la recherche, ouvrant la voie à des progrès majeurs en médecine génétique personnalisée. La recherche est publiée dans le American Journal of Human Genetics.
Les maladies complexes comme le cancer de Parkinson et le sein et les conditions comme le cholestérol élevé ne sont pas les mêmes pour tous ceux qui en ont. Bien que les patients puissent partager le même diagnostic, le chemin qui les a conduit peut différer considérablement; Certains cas peuvent être causés par l’effet combiné de milliers de variations génétiques courantes, tandis que d’autres peuvent être entraînées en grande partie par une seule mutation rare et nocive dans un gène important. Jusqu’à présent, les études génétiques à grande échelle ont eu tendance à brouiller ces différences, faisant la moyenne des effets génétiques de tous les patients.
« Tout le monde atteint une maladie complexe n’arrive pas de la même manière », a déclaré l’auteur principal Chad Shaw, statisticien avec des nominations conjointes à Rice et Baylor et membre du corps professoral du NRI. Shaw est également le directeur de Rice’s Data to Knowledge Lab.
« Le pivot causal est conçu pour détecter ces différences et trier les patients en sous-groupes plus précis et biologiquement significatifs. Il s’agit d’une étape fondamentale vers une médecine génétique vraiment personnalisée. »
L’étape est une nouvelle extension de la méthodologie de l’analyse causale, qui est un domaine de la science statistique que Shaw enseigne au riz.
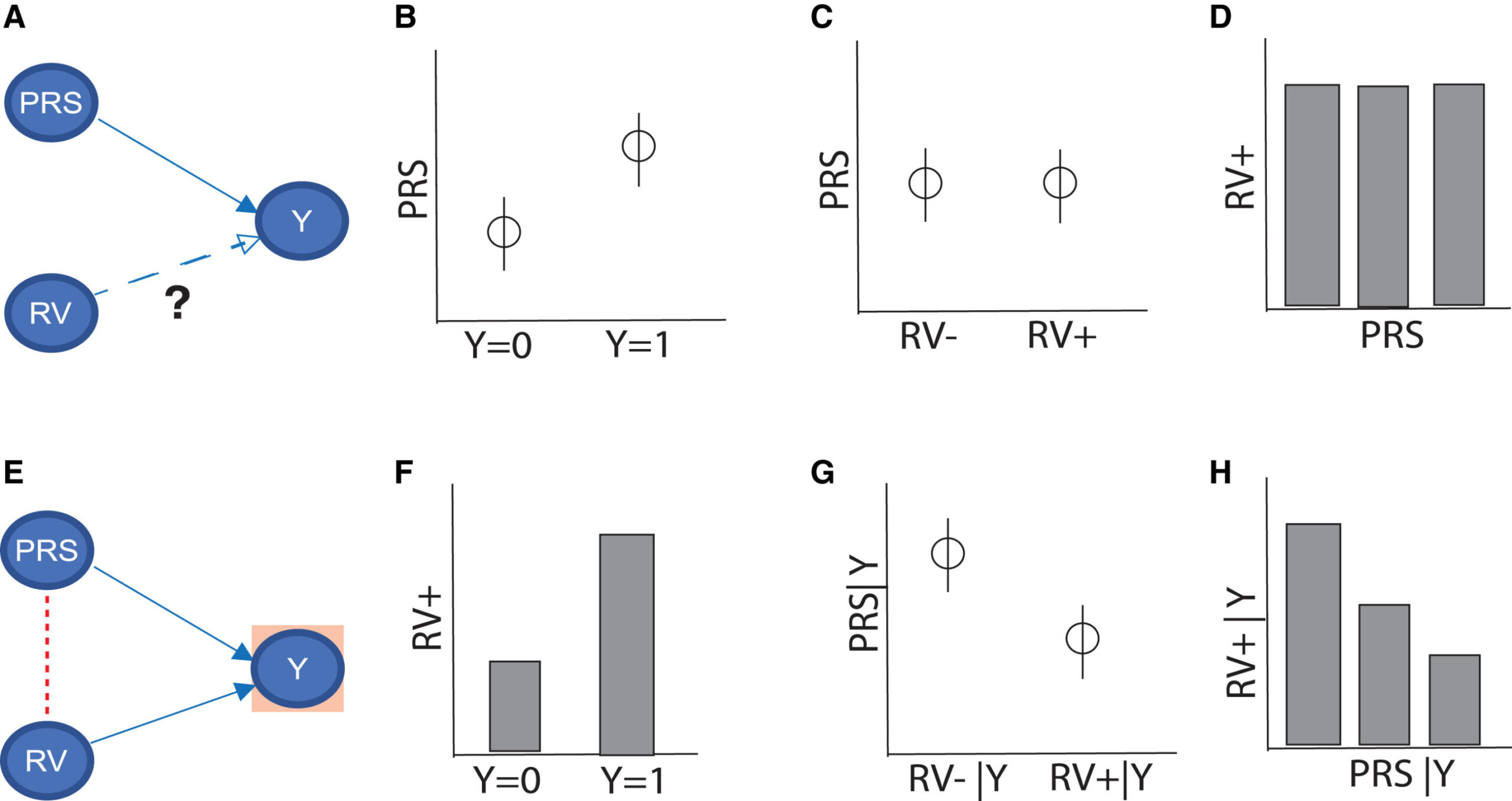
Le pivot causal exploite quelque chose appelé un score de risque polygénique (PRS), qui est un résumé de l’effet combiné de nombreuses variantes génétiques courantes et de quelque chose que les chercheurs connaissent souvent déjà sur de nombreuses maladies. Le PRS agit comme un «pivot» par rapport à lequel la méthode teste d’autres causes potentielles telles que des changements d’ADN rares et nocifs appelés variantes rares.
Si une variante rare entraîne vraiment des maladies chez certaines personnes, alors parmi les patients atteints de maladie, ceux qui portent la variante ont tendance à avoir des nombres PRS inférieurs à ceux qui ne le font pas (parce que la variante rare elle-même fournit la poussée dans la maladie). Le pivot causal formalise ce concept en un test statistique rigoureux qui peut identifier ces rares sous-groupes axés sur les variants et estimer la taille de leur effet.
Contrairement aux études d’association traditionnelles à l’échelle du génome, qui nécessitent de grands ensembles de données de contrôle et de contrôle, le pivot causal peut fonctionner dans des conceptions uniquement – un avantage dans des contextes réels où des échantillons de contrôle sain peuvent être indisponibles, comme dans des sous-types rares, certains essais cliniques ou études sur les réactions médicamenteuses négatives. L’équipe a également développé des garanties contre des facteurs de confusion comme l’ascendance, garantissant la fiabilité de la méthode dans diverses populations.
En utilisant les données de la biobanque britannique, qui comprend des informations génétiques et de santé de plus d’un demi-million de volontaires, les chercheurs ont validé la méthode sur trois paires de réglementation des gènes bien étudiées: variantes LDLR chez les personnes avec un cholestérol LDL très élevé (hypercholestérolémie); Variantes BRCA1 dans le cancer du sein; et les variantes GBA1 dans la maladie de Parkinson.
Dans chaque cas, le pivot causal a détecté des signaux clairs conformes à la biologie connue – preuve approfondie que la méthode fonctionne comme prévu. Les tests croisés (comme la recherche de l’effet de GBA1 dans le cancer du sein) n’ont montré aucun signal, et des variantes « synonymes » inoffensives n’ont produit aucun faux point positif.
L’équipe est allée plus loin dans les tests de Parkinson, appliquant l’approche sur une voie de stockage lysosomale, qui est un groupe de gènes impliqués dans le recyclage des déchets cellulaires. Ils ont constaté que les patients présentant un fardeau plus lourd de variantes rares dans cette voie avaient tendance à avoir des nombres PRS inférieurs, suggérant que de multiples hits rares peuvent se combiner pour créer une voie alternative vers la maladie.
« Ce type de détection de sous-groupe change le jeu », a déclaré Shaw. « Il ouvre la porte aux tests cliniques et aux outils qui peuvent faire correspondre les patients aux thérapies en fonction du mécanisme réel qui stimule leur maladie, pas seulement le nom de la maladie. »
Les implications pour la médecine personnalisée sont d’une grande portée. En révélant les «routes» génétiques vers une maladie, le pivot causal pourrait améliorer les stratégies de test génétique, aidant les médecins à décider qui devrait être testé pour des mutations rares spécifiques. Il pourrait également permettre des essais cliniques plus ciblés, en se concentrant sur les patients les plus susceptibles de répondre à une thérapie donnée. Encore plus, il fournit un cadre pour classer les patients par mécanisme et pas seulement les symptômes, ouvrant la voie à des traitements spécifiques au mécanisme.
« La médecine personnalisée sera fondée sur un séquençage complet du génome pour chaque patient, mais il est difficile pour les prestataires de soins de santé de comprendre car il y a plus de 5 millions de variantes dans le génome de chaque personne », a déclaré le Dr John Belmont, professeur adjoint de génétique moléculaire et humaine au Baylor College of Medicine et l’un des auteurs de l’article.
« Les workflows d’interprétation actuels sont basés sur les règles, non calibrés et ne produisent pas de probabilités causales au niveau du patient. L’analyse causale, en mettant l’accent sur les effets des interventions, est idéale pour la prise de décision médicale. Le nouveau travail du Dr Shaw est un pas sur la route pour intégrer officiellement les informations des cohortes cliniques et de la population.
« La plupart des études génétiques des maladies humaines se sont concentrées exclusivement sur les variations génétiques courantes ou rares – les changements dans le code de l’ADN », a déclaré le co-auteur, le Dr Joshua Shulman, professeur de neurologie, de neuroscience et de génétique moléculaire et humaine à Baylor et co-réalisateur du NRI.
« L’équipe du Dr Shaw a créé un modèle statistique élégant qui aide à unifier ces approches, permettant une analyse plus puissante et intégrée. Nous sommes ravis d’utiliser cette stratégie pour découvrir de nouveaux facteurs de risque génétiques pour la maladie d’Alzheimer et de Parkinson. »
Les chercheurs disent qu’ils envisagent de futures applications qui vont au-delà de la génétique. À la place du PRS, le pivot pourrait être n’importe quel conducteur de maladie établi – une exposition environnementale, un biomarqueur ou même une caractéristique d’imagerie, par exemple – faire du cadre un moteur de découverte polyvalent pour plusieurs branches de médecine.
« La médecine personnalisée a besoin de structure », a déclaré Shaw. « Nous avons créé un moyen clair et testable de tenir compte de la diversité génétique dans une maladie. C’est un outil que nous pouvons utiliser sur de grands ensembles de données aujourd’hui, et nous pouvons nous adapter à la clinique demain. »
Cette étude collaborative comprenait CJ Williams chez Genetics & Genomics Services Inc.; Taotao Tan à Baylor; et Daniel Illera et Nicholas Di à Rice.