Le séquençage de l’ARN est apparu comme un supplément puissant du séquençage de l’ADN pour le diagnostic de la maladie mendélienne, mais la traduction clinique de l’ARN-seq diagnostique n’a pas été largement atteinte. Des chercheurs du laboratoire de génétique médicale et multioomique du Baylor College of Medicine ont publié la validation clinique du premier test de séquençage d’ARN pour l’analyse diagnostique du transcriptome entier pour les troubles génétiques.
Les résultats, qui apparaissent dans The American Journal of Human Geneticsétablir un pipeline de séquençage de transcriptome complet à usage clinique, élargissant le rôle du séquençage d’ARN au-delà de l’analyse ciblée.
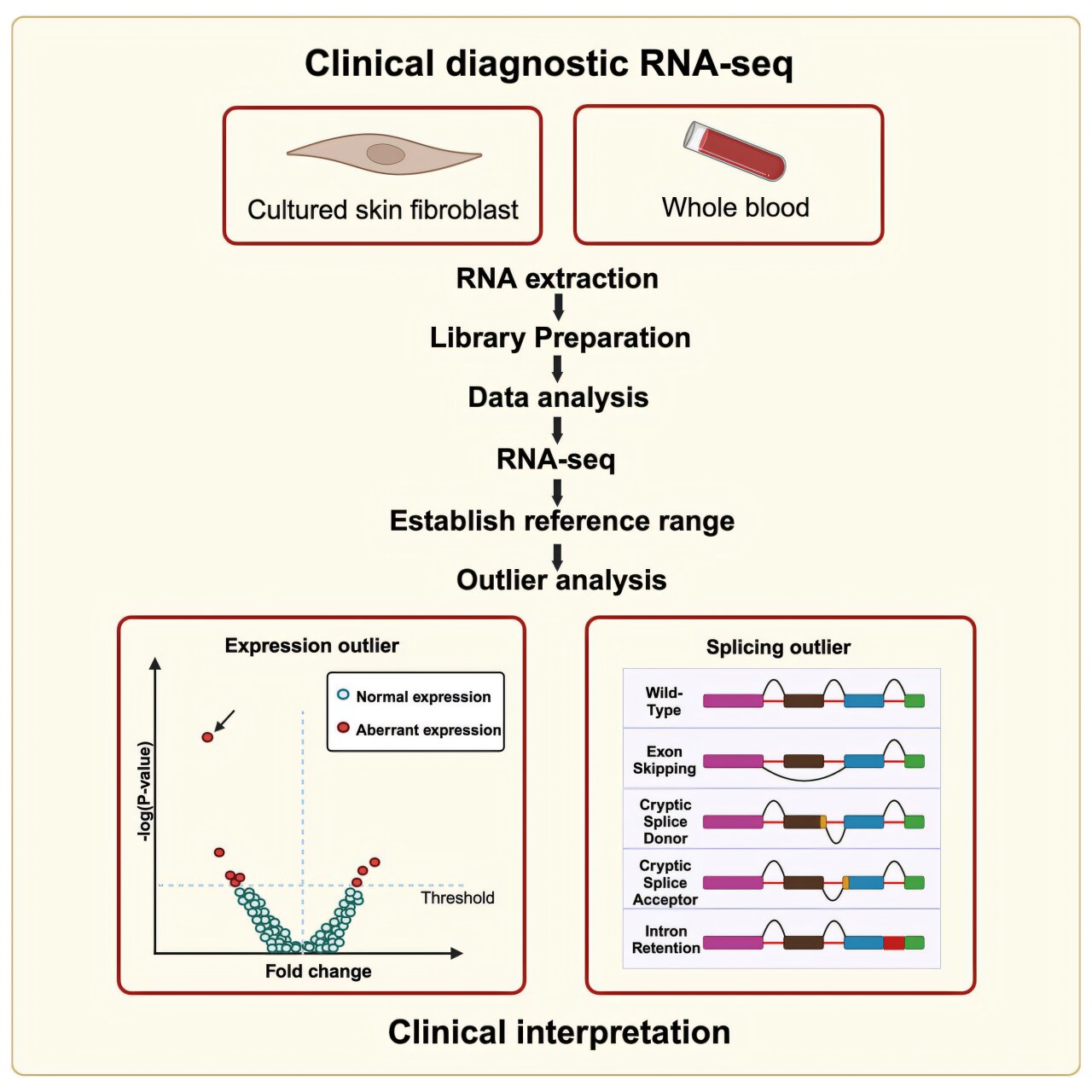
« Cette validation représente la première tentative de libérer le potentiel de transcriptome complet du séquençage de l’ARN pour les diagnostics cliniques », a déclaré l’auteur correspondant, le Dr Pengfei Liu, professeur agrégé de génétique moléculaire et humaine et directeur du laboratoire médical génétique et multiomique (MGML) à Baylor. « Bien que le séquençage de l’ARN ait été utilisé dans les tests cliniques, il a été limité à l’analyse des gènes ciblée. En fait, lorsque nous avons demandé l’accréditation du CAP (Collège des pathologistes américains), nous avons travaillé avec eux pour créer la première activité de séquençage en transcriptome entièrement en tant que test. »
Le test RNA-Seq utilise des échantillons à partir de fibroblastes ou de sang et d’analyses pour les valeurs aberrantes dans l’expression des gènes et les modèles d’épissage. Les chercheurs ont validé le test en utilisant 130 échantillons, dont 40 échantillons avec un diagnostic moléculaire positif et 90 échantillons négatifs de personnes apparemment en bonne santé pour le contrôle. Ils ont développé des repères pour l’expression et l’épissage en utilisant des données RNA-Seq à partir d’un échantillon lymphoblastoïde dans le génome accessible au public dans un consortium de bouteilles. L’équipe a établi des plages de référence pour chaque gène en fonction des données de contrôle.
Pour évaluer les performances cliniques, l’équipe a évalué le test en utilisant des échantillons avec des résultats de diagnostic précédemment identifiés du réseau de maladies non diagnostiqué (UDN). L’étude a confirmé la capacité du test à détecter les résultats de l’ARN diagnostique à travers les modes d’analyse axés sur le transcriptome et à ADN, démontrant son potentiel pour améliorer le diagnostic moléculaire.
Ce test est le premier lancé par le MGML et est maintenant proposé pour le séquençage clinique de l’ARN dans l’UDN.