Les médecins traitant une maladie rénale dépendent depuis longtemps des essais et des erreurs pour trouver les meilleures thérapies pour les patients individuels. Désormais, de nouveaux outils d’intelligence artificielle (IA) développés par des chercheurs de la Perelman School of Medicine et de la Wharton School de l’Université de Pennsylvanie peuvent analyser les maladies rénales au niveau cellulaire pour correspondre aux traitements les plus efficaces et accélérer les solutions. Cette percée, publiée cette semaine Génétique de la naturepourrait avoir un impact sur des millions de personnes atteintes d’une maladie rénale.
« Nous passons de la conjecture à la précision », a déclaré Katalin Susztak, MD, Ph.D., professeur de néphrologie, génétique et directeur du Penn / Chop Kidney Innovation Center. « Les maladies rénales ne sont pas identiques, mais l’utilisation de l’IA nous a aidés à identifier et à cataloguer 70 types distincts de cellules rénales qui apparaissent à travers des échantillons humains et animaux. Cela améliore la fiabilité de la recherche et peut conduire à des traitements potentiels. »
Les outils d’IA transforment les soins rénaux
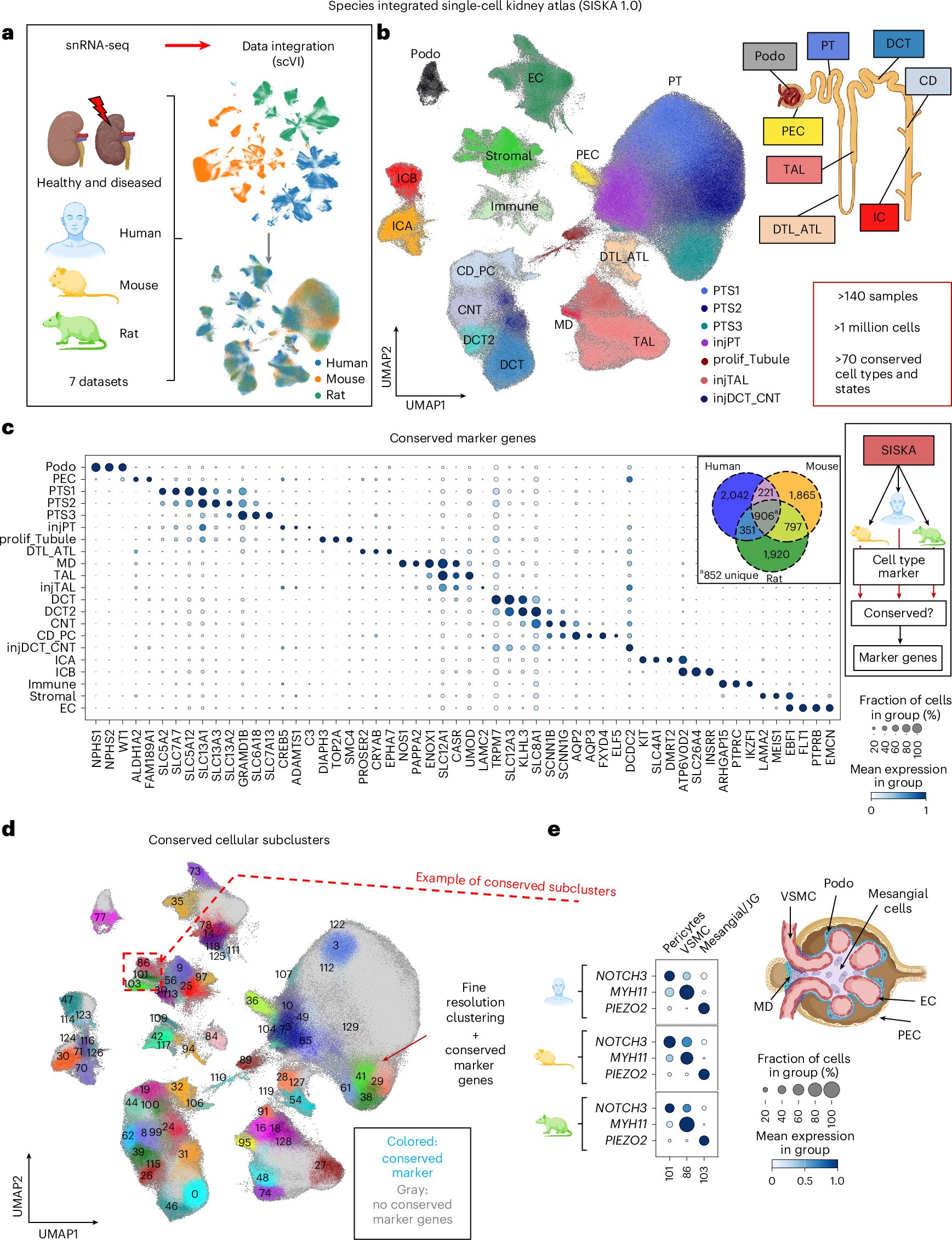
L’équipe de Penn a relevé un défi majeur dans le séquençage de l’ARN à cellule, une technique de pointe qui examine l’activité génétique des cellules individuelles. Jusqu’à présent, cette méthode a été difficile à appliquer aux patients individuels en raison de la définition de type de cellule incohérente et de l’incertitude sur les modèles de laboratoire (comme les souris ou les rats) les meilleures maladies humaines.
La solution de l’équipe comprend Siska 1.0 Atlas: un ensemble de données massif construit à partir de plus d’un million de cellules à travers 140 échantillons de rein humains, de souris et de rat. En combinaison avec une nouvelle méthode statistique qui examine les programmes génétiques – des ensembles de gènes co-régulés représentant des voies biologiques – plutôt que des gènes individuels, il était plus facile de repérer des problèmes liés à la maladie dans les cellules d’une personne. Le nouvel outil open-source, appelé CellSpectra, a été créé directement à Penn.
« Nous avons construit CellSpectra pour faire ce que les méthodes actuelles ne peuvent pas: analyser l’échantillon d’un patient à la fois et l’interpréter dans le contexte des espèces, des maladies et de la thérapie », a déclaré Nancy Zhang, Ph.D., professeur de statistique et de science des données de Ge Li et Ning Zhao à l’école Wharton.
« Ces deux outils seront gratuits pour quiconque peut utiliser. Désormais, les chercheurs, les scientifiques et les cliniciens auront tous accès à ces outils qui permettent des traitements personnalisés avec une plus grande précision », a ajouté Susztak.
Au-delà de l’ARN: un nouveau catalogue de protéines et une carte de traits de maladie
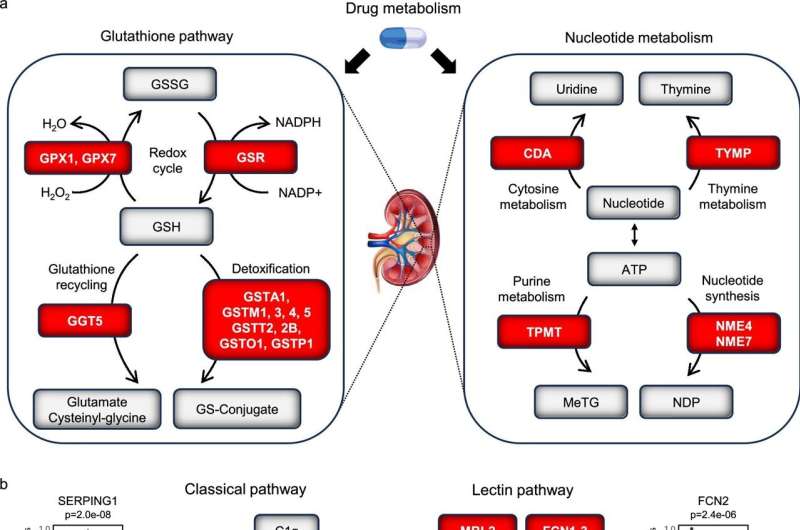
Dans une étude distincte, l’équipe de Susztak a également créé le premier catalogue complet de protéines rénales, offrant une nouvelle lentille sur la façon dont l’abondance des protéines, pas seulement l’expression des gènes, contribue à la maladie. Cette œuvre, publiée dans Médecine de la naturea constaté que les niveaux de protéines dans les cellules rénales ne correspondent souvent pas à l’activité des gènes (un décalage appelé discordance), montrant que l’étude des gènes seuls ne suffit pas pour comprendre comment les maladies se développent.
« Il s’agit d’un pas en avant significatif dans la compréhension de la biologie de la maladie rénale – pas seulement au niveau de l’ARN, mais aussi au niveau des protéines fonctionnelles », a déclaré Susztak. « Lier les profils de protéines avec des traits tels que la pression artérielle, les niveaux de lipides et la fonction rénale ouvre de nouvelles portes pour les thérapies qui ciblent les bonnes molécules chez les bons patients. »